清华团队定位新冠病毒和人受体作用位点 助力抗体筛选
导读:新冠病毒如何进入人体?近日,国内外多家实验室试图从近原子层面破解谜团。
新冠病毒如何进入人体?近日,国内外多家实验室试图从近原子层面破解谜团。当地时间2月19日,清华大学生命学院王新泉课题组和医学院张林琦课题组在生命科学预印本网站BioRxiv(未经同行评议)发表论文,利用X射线衍射技术,解析了新冠病毒表面刺突蛋白受体结合区(RBD)与人受体血管紧张素转化酶2(ACE2)复合物的晶体结构。
该团队获得的复合物结构分辨率为2.45埃,是截至目前多家实验室中获得的最高分辨率。研究团队准确定位出新冠病毒RBD和受体ACE2的相互作用位点。
病毒要进入人体细胞,必须找到人体细胞上相应的受体蛋白。因此受体就好比是一把“锁”,得有相应的“钥匙”才能打开,而后病毒才能进入细胞内部。新冠病毒针对ACE2受体的“钥匙”就是刺突蛋白,即S蛋白。
这项最新的研究发现,新冠病毒在关键的受体结合氨基酸位点与SARS病毒大同小异。基于深入的对比分析,研究团队还发现了一些可能造成新冠病毒与SARS病毒传播差异的氨基酸位点,以及导致针对SARS病毒的抗体不能够有效抑制新冠病毒感染的氨基酸位点。
王新泉与张林琦实验室在新发与再发病毒感染的分子机制、中和抗体筛选和鉴定、疫苗开发等领域开展合作近10年。此前曾进行中东呼吸道综合征冠状病毒(MERS-CoV)抗体MERS-4抗病毒机制的研究,他们合作取得了一系列国际前沿性的研究成果。
具体来说,研究团队利用昆虫细胞体系表达和纯化了新冠病毒 RBD和人ACE2胞外结构域,成功生长出新冠病毒 RBD-ACE2复合物的晶体(晶体生长条件:100 mM MES, pH 6.5, 10% PEG5000mme, 12% 1-propanol)。此后,他们采用X射线晶体学,利用中国科学院上海光源(SSRF)BL17U线站收集了分辨率为2.45埃的衍射数据,并解析了这一复合物的三维空间结构。
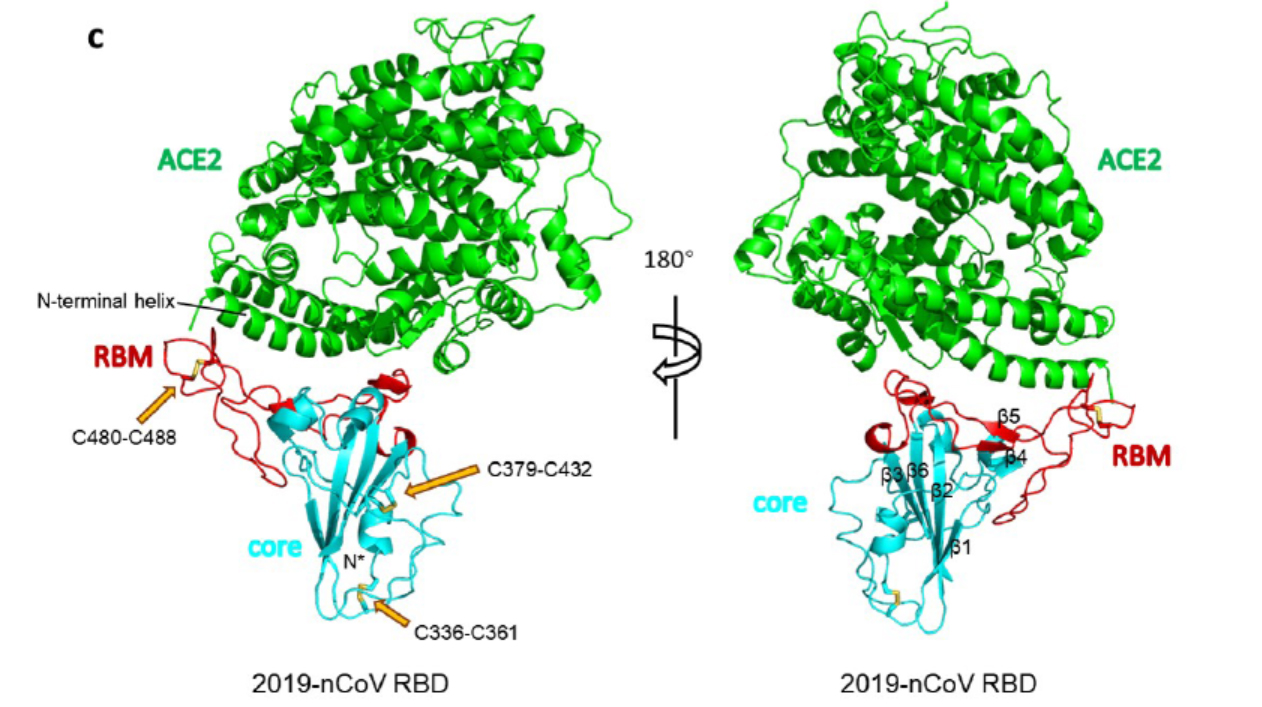
与ACE2结合的2019-nCoV RBD的总体结构:ACE2为绿色,2019-nCoV RBD核心为青色,RBM为红色,2019-nCoV RBD中的二硫键显示为棒状并用黄色箭头指示,负责结合的ACE2的N末端螺旋被标记
值得一提的是,此前美国德克萨斯大学奥斯汀分校Jason S. McLellan研究组在《科学》(Science)杂志正式发表论文,他们利用冷冻电镜技术率先解析了2019-nCoV表面S蛋白的近原子结构。
但是,已有的模型中RBD的分辨率不完全,特别是对于与ACE2直接产生相互作用的受体结合模体(RBM)。研究团队认为,对2019-CoV RBD和ACE2之间相互作用的计算机建模已经确定了一些可能参与实际相互作用的氨基酸残基,但作用的过程与细节仍然难以捉摸。
研究团队此次结构解析得出,2019-nCoV RBD / ACE2和SARS-CoV RBD / ACE2界面在掩埋表面积,相互作用残基数和亲水相互作用网络方面都具有实质上的相似性,但在表面静电势上存在一些差异。
尽管这两种病毒处于乙型冠状病毒属的不同遗传谱系中,但研究者认为,这种结构和序列上的相似性有力地证明,2019-nCoV和SARS-CoV的RBD存在着趋同进化,从而提高它们与相同的ACE2受体的结合亲和力。
团队还认为,既然结构具有相似性,那么ACE2与2019-nCoV和SARS-CoV的RBD之间的结合亲和力也在先前报道的相同范围内(约为10-60 nM)。
而针对近日McLellan研究组得出的“新冠病毒S蛋白与ACE2的亲和力,是SARS病毒S蛋白与ACE2之间亲和力10至20倍”的结论,作者们认为,这可能是由于分析中使用的蛋白质不同,或其他一些未知原因。
事实上,作者们认为,仅考虑结合的亲和力强弱不太能解释2019-nCoV异常强的传染性。其他因素,例如2019-nCoV刺突蛋白S1/S2边界处特有的“ RRAR”弗林蛋白酶切位点,可能在促使其人与人之间快速传播上起着更重要的作用。
值得一提的是,中和抗体被视为免疫系统对抗病毒感染的策略之一。据此前的研究,2019-nCoV可以被马的抗SARS-CoV血清和来自恢复期SARS患者的血清交叉中和,这也进一步强调了2019-nCoV与SARS-CoV RBDs的结构相似性。
然而,论文提到,迄今为止,针对SARS-CoV的抗体(m396, S230, 80R和CR3014)还没有显示出对2019-nCoV S蛋白或RBD有理想的交叉结合和中和活性。
唯一的例外是SARS-CoV特异性人类单克隆抗体CR3022,该抗体可以与2019-nCoV RBD有效结合。不过,目前尚不确定CR3022在SARS-CoV或2019-nCoV RBDs上的确切抗原表位。
在不能与2019-nCoV RBD结合的3种抗体中,研究团队通过对SARS-CoV RBD-Fab复合物的高分辨晶体结构解析了其中2种(m396和80R)的表位。通过将这些表位残基映射到SARS-CoV RBD序列和2019-nCoV RBD序列,研究团队发现,和SARS-CoV相比,2019-nCoV RBD上抗体m396的21个抗原表位有7个残基改变,抗体80R的24个抗原表位则有15个残基改变。
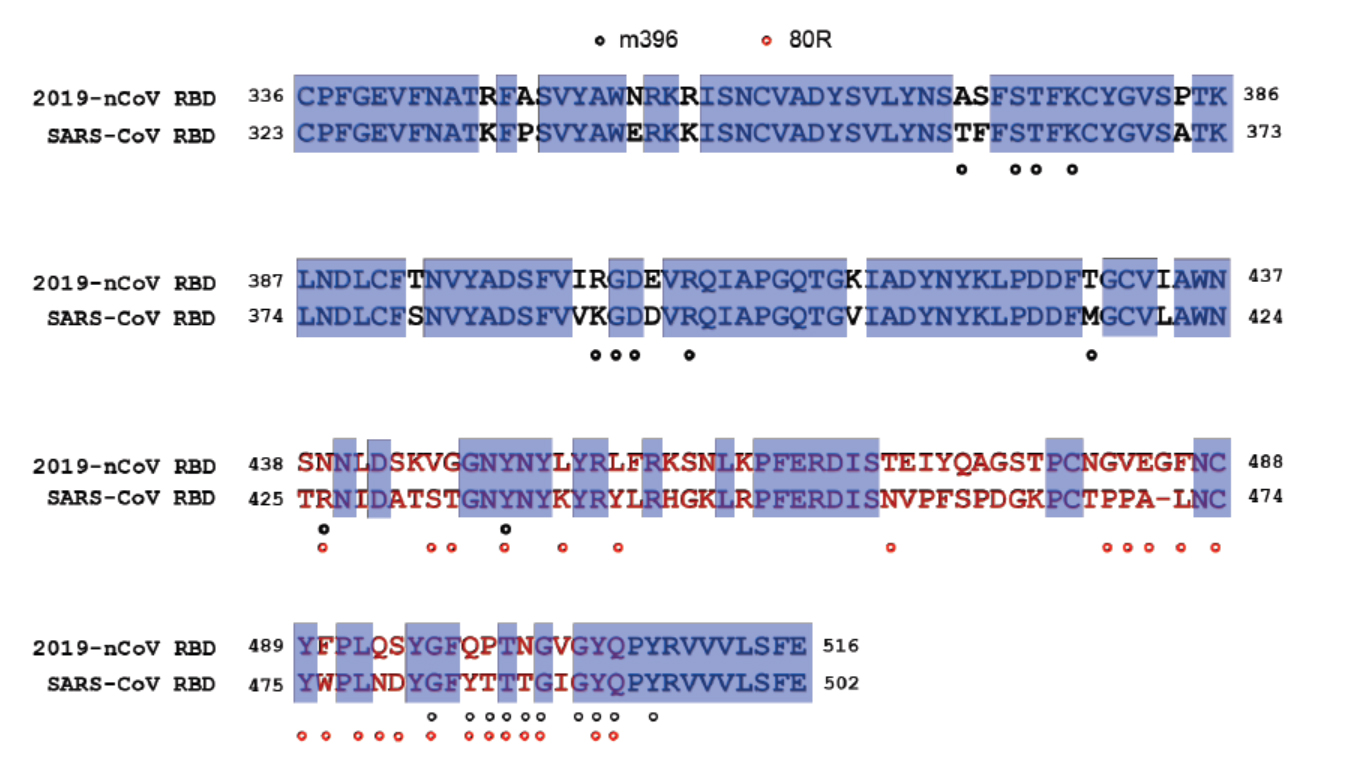
SARS-CoV中和抗体抗原表位映射:黑点表示m396的表位残基;红点表示80R的表位残基。
论文提到,这可能为m396和80R对2019-nCoV没有显示交叉反应提供了结构基础。然而,2019-nCoV RBD和SARS-nCoV RBD之间确实存在氨基酸保守性,甚至在更多变的RBM中也存在(上图)。
研究团队认为,这项研究为治疗性抗体药物开发以及疫苗的设计奠定了坚实的基础。张林琦表示,现在疫苗研发的关键靶点就是针对新冠病毒的“钥匙”展开的,病毒如何进入细胞这一步非常关键。这些研究将有利于科学家筛选能够阻止病毒与人体结合的抗体或小分子药物。两个团队下一步的工作重点是基于结构设计筛选能够阻止RBD与ACE2结合的抗体或者小分子药物。
但他们同样强调,这是一个相对漫长的过程,因为迄今为止能够有效抑制新冠病毒的特异性抗体和药物都还在筛选和验证过程中,这需要更多科学家不断的努力。
值得一提的是,此前的2月17日,西湖大学周强教授团队在全球范围内率先报道了ACE2全长蛋白的高分辨电镜结构(2.9埃),2月19日,该团队进一步解析出ACE2全长蛋白与新冠病毒S蛋白RBD的复合物电镜结构(3.5埃)。此外,中科院微生物研究所齐建勋团队也解析了新冠病毒RBD与ACE2复合物的晶体结构(2.5埃)。
上述提及的国内三个独立团队都选择在第一时间将其复合物的原子坐标向全社会公布,以提高它们的利用率。